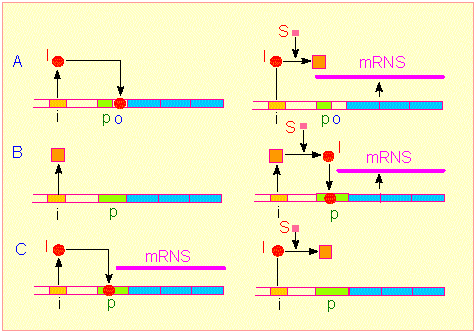
Man unterscheidet zwischen Regulatorgenen (ocker) und Strukturgenen (hellblaz). Einer Gruppe von Strukturgenen (einem Operon) ist ein Promotor (grün: Startstelle der DNS-abhängigen RNS-Polymerase) und ein Operator (o: Bindungsstelle eines Regulatormoleküls [Induktor oder Repressor]) vorgeschaltet. Oberes Bildpaar: Substratinduzierte Kontrolle der Transkription. Der vom Regulatorgen codierte Repressor bindet an o und verhindert damit die Transkription der Strukturgene. Er ist durch ein Substrat inaktivierbar, damit wird die Transkription (gebildete RNS: violette Linie) der Strukturgene freigegeben. Die durch die Strukturgene codierten Enzyme sind am Abbau des genannten Substrats beteiligt (JACOB-MONOD-Modell). Mittleres Bildpaar: Vom Regulatorgen wird ein inaktiver Induktor codiert. Die Transkriptionsrate der Strukturgene ist gering. Durch ein Substrat wird der Induktor aktiviert, er bindet an den Promotor und fördert damit die Aktivität der DNS-abhängigen RNS-Polymerase, die Transkriptionsrate steigt. Unteres Bildpaar: Vom Regulatorgen wird ein aktiver Induktor codiert, die Transkriptionsrate der Strukturgene ist hoch. Durch Bindung eines Substratmoleküls wird der Induktor inaktiviert.