Für funktionsoptimierte Gebrauchsgegenstände gilt form follows function. Wenn ein Proteinmolekül funktionieren soll, muß es auch eine dafür geeignete Form haben. Hier bedingt jedoch die sich aus der Evolution ergebende Form der Aminosäurekette die mögliche Funktion als Enzym, Strukturprotein oder Effektor. Die Entwicklung der Natur hat bisher ohne menschliches Zutun für Strukturen und daraus resultierende Funktionen gesorgt. Für Eigenentwicklungen ("protein engineering") auf der Basis herkömmlicher Proteine muß der Mensch zunächst einmal den Zusammenhang von Proteinstruktur und Funktion kennen lernen. Dabei ist die bildliche Darstellung von Strukturen extrem hilfreich. Einige Werkzeuge zur Verdeutlichung von Proteinstrukturen sollen hier gezeigt werden.
Am Anfang steht die Struktur selber - nur, wenn die räumliche Lage der Atome eines Moleküls zueinander bekannt ist, kann auch etwas dargestellt werden. In der Regel werden die Atomkoordinaten aus den Beugungsbildern von Röntgenstrahlen an Proteinkristallen gewonnen. Listen mit den Koordinaten werden zentral in einer Datenbank abgelegt (PDB; Protein Data Bank, USA). Auf elektronischem Weg können die Daten von dort erhalten werden (http://www.rcsb.org). In anderen Erdteilen werden die Daten ebenfalls zur Verfügung gestellt, z.B. http://www.embl-heidelberg.de/srs5/. Im Januar 1998 enthielt die PDB die Daten zu 6419 Proteinen, Peptiden und Viren, 100 Protein/Nukleinsäure-Komplexen, 516 Nukleinsäuren und 12 Kohlehydraten.
Im Zeitalter der Computergraphik mag sich wohl niemand mehr die Mühe machen, ein den Atomkoordinaten folgendes Drahtmodell eines Proteins zu biegen oder riesige Kalottenmodelle zusammenzustecken. In den heutigen Computerprogrammen sind jedoch noch Elemente aus der "von Hand"-Zeit zu finden. Hier sollen die Programme RasMol (1) und MolScript (2) gezeigt werden, mit denen einige Bilder für dieses Projekt erstellt wurden. RasMol ist in Versionen für verschiedene Rechnerplattformen im Internet erhältlich (z. B. für DOS-Rechner vom EMBL Anonymous FTP file server: ftp.embl-heidelberg.de/pub/software/dos/ oder über die eigene homepage http://www.umass.edu/microbio/rasmol/). Für Molscript muß mit dem Autor ein Lizenzvertrag geschlossen werden (http://www.avatar.se/molscript/).
Wie sieht ein Proteinmolekül aus? Ein benachbartes Molekül sieht nur die Elektronenwolke, nicht aber die darunterliegenden Atomkerne. Für das menschliche Verständnis der Abhängigkeit der Funktion von strukturbildenden Aminosäuren ist der Anblick einer diffusen Wolke jedoch nicht ausreichend. Um Strukturmerkmale darzustellen, gibt es verschiedene Stufen der Abstraktion von den "wirklichen" Verhältnissen. Für jedes Veranschaulichungsproblem muß ein Kompromiß zwischen Durchsichtigkeit und Kompaktheit des Moleküls gefunden werden. Außer den Formen der Abbildungselemente wird die Farbe als Gestaltungselement benutzt.
RasMol ist ein "automatisches" Visualisierungsprogramm. Es liest den Datensatz mit den Atomkoordinaten eines Makromoleküls sowie eventuell enthaltene Zusatzinformationen. Der räumliche Verlauf der Aminosäurekette kann auf verschiedene Arten symbolisiert werden:
Die folgenden Bildbeispiele zeigen eine Protein-Untereinheit des Tabakmosaikvirus (TMV) zusammen mit einem gebundenen Trinucleotid aus dem RNA-Genom. Wie die Farbgebung zur Verdeutlichung der Strukturmerkmale benutzt wurde, ist in den Bildlegenden erläutert. Die Abbildungen wurden mit RasMol Version 2.2 erstellt. Die aktuelle Version ist 2.6 mit einer Reihe zusätzlicher Funktionen; Auskunft darüber gibt es über die RasMol-Homepage.
Die einfachste Darstellung ist eine Verbindung der C-alpha-Atome der einzelnen Aminosäuren (backbone). Das Molekül ist in dieser Darstellung sehr durchsichtig, Sekundärstruktur läßt sich erahnen. Der Gesamteindruck der Raumausfüllung fehlt wegen der nicht abgebildeten Seitenketten der Aminosäuren.
![[kga.htm]](../fo17_1/kgai.gif)
Die räumliche Anordnung der Proteindomänen wird erst durch Drehen des Moleküls auf dem Bildschirm klar.
Ein besserer Eindruck von der Raumfülle entsteht, wenn alle chemischen Bindungen zwischen den Atomen der Aminosäuren wie in einem Drahtmodell gezeigt werden (wire frame). Bei aller Durchsichtigkeit, die diese filigrane Darstellung bietet, erkennt man gut die Komplexität eines Proteins, sieht aber "den Wald vor Bäumen" nicht.
![[wha.htm]](../fo17_1/whai.gif)
In dem Modus "sticks" wird die Darstellung wegen massiverer Bindungsdarstellung kompakter. Bei "ball and sticks" wird außerdem jedes Atom durch eine Kugel symbolisiert, sodaß ein Eindruck der Masseverteilung innerhalb des Moleküls entsteht.
![[ssa.htm]](../fo17_1/ssai.gif)
![[bca.htm]](../fo17_1/bcai.gif) Ball & sticks (18 k)
Ball & sticks (18 k)Im Hintergrund liegende Teile werden zunehmend verdeckt und können erst durch Drehung der Struktur sichtbar gemacht werden.
Einen Eindruck von der Elektronenwolke im Bereich der Wirkung der Van der Waals-Kräfte erhält man durch die Ansicht im "space fill"-Modus. Hierbei erkennt man nur noch die dem Betrachter zugewandte Oberfläche des Moleküls, es ist vollkommen undurchsichtig. Diese Darstellung entspricht den herkömmlichen Kalottenmodellen.
![[pha.htm]](../fo17_1/phai.gif)
Die intramolekularen Wechselwirkungen der Aminosäuren bestimmen die Sekundärstrukturelemente eines Proteins. Die wichtigsten Elemente sind regulär gewundene Bereiche (Helices), zu Faltblättern aneinandergelegte beta-Stränge, durch Wasserstoffbrücken gehaltene Knicke (bends, turns) sowie die ungeordneten Coils. Zur Verdeutlichung dieser Elemente gibt es den Darstellungsmodus "ribbons", in dem von dem tatsächlichen Verlauf der C-alpha-Atomverbindungen so weit abstrahiert wird, daß der Aminosäurekettenverlauf als spiralig gewundenes Band in Helices oder flachgestrecktes Band in Faltblättern abgebildet wird. Die anderen Bereiche werden als geschwungene Linien dargestellt, die nur noch die relative Lage der Aminosäurekette andeuten.
![[rta.htm]](../fo17_1/rtai.gif)
Mit RasMol ist bei geeigneter Auswahl der hier vorgestellten Bildschirmparameter schnell ein aussagekräftiger Überblick zu einem PDB-Datensatz zu erhalten. Bedient man sich der Scriptsprache des Programmes, können die beschriebenen Gestaltungsmerkmale auch für Teile der Makromoleküe verwendet werden (Hervorhebung einzelner Aminosäuren etc.).
Für Demonstationen im WorldWideWeb ist RasMol weiterentwickelt worden. Mit dem für Netscape erhältlichen Zusatzprogramm Chime können vom Betrachter steuerbare "Filme" angesehen werden. Das Programm und Beispiele sind über die Chime-Homepage im Internet zugänglich. Einige Beispiele für makromolekulare Strukturen finden sich über den FB Biologie der Universität Hamburg. Hier TMV als Beispiel. Auch kleine Moleküle sind damit leicht räumlich zu veranschaulichen, wie in den Chemie-Kapiteln von Botanik online.
Chime wird auch von Datenbankanbietern zur Darstellung von Molekülen eingesetzt. Wenn z. B. PDB-Daten über den 3DB Browser gesucht werden, erscheinen unter dem Suchergebnis in der Rubrik Visual 3D analysis Links zu den Programmen STING und GRASS, die sofort einen Überblick über die Moleküleigenschaften geben.
Zur Darstellung von Details einer Makromolekülstruktur in druckreifer Qualität ist das Programm MolScript besser geeignet, da es noch differenziertere Möglichkeiten bietet. Allerdings arbeitet es nicht automatisch, der Benutzer muß alle gewünschten Features von Hand in der dem Programm eigenen Sprache angeben. Vor allen Dingen muß dem Benutzer bekannt sein, welche Strukturelemente wo in dem Molekül lokalisiert sind. Diese Informationen sind nur in einem Teil der PDB-Einträge enthalten. Es gibt jedoch eine andere Datenbank, DSSP (3), die für ausgewählte Proteine die Bindungsdaten aller Wasserstoffbrücken sowie ausführlich alle Strukturmerkmale auflistet. DSSP-Daten sind ebenfalls über Brookhaven (oder embl) erhältlich. Die Darstellung von Strukturelementen mit MolScript orientiert sich an Regeln, die von J. S. Richardson für die graphische Präsentation von Proteinen erstellt wurden (4). Das Rechenergebnis von MolScript ist ein PostScript-File. Der Inhalt dieses Files kann entweder auf einem entsprechenden Drucker zu Papier gebracht oder mit anderer Software (z. B. GhostScript) auf einem Bildschirm sichtbar gemacht werden.
Die Darstellungsmöglichkeiten mit MolScript sind so vielfältig, daß hier nicht alle Punkte aufgezählt werden können. Jeder einzelnen Aminosäure (oder Gruppe von Aminosäuren) müssen zur Darstellung Formattribute zugeordnet werden. Farbe kann ebenfalls für Atome, Aminosäuren oder Gruppen oder aber für ganze Moleküle angegeben werden. Als Beispiel dient wieder das TMV-Protein.
![[2tmva.gif]](../fo17_1/2tmvai.gif) (9 k) | (104 k, ab Netscape2)
(9 k) | (104 k, ab Netscape2)
![[2tmva.gif]](../fo17_1/2tmvai.gif)
Ein anderes Beispiel ist die Darstellung der NAD-Bindungsstelle in der Lactatdehydrogenase. Dort wurden außer den globalen Merkmalen Helix und beta-Strang die an der Bindung des Coenzyms beteiligten Aminosäuren hervorgehoben: Die Atome der über Wasserstoffbrücken bindenden Aminosäuren sind als farbige Kugeln dargestellt, die Atome der Aminosäuren, die für die Bindung eine "hydrophobe Tasche" bilden, sind durch weiße Kugeln abgebildet.
![[2ldba.gif]](../fo17_1/2ldbai.gif)
Zur besseren Darstellung der dreidimensionalen Struktur können sehr einfach Stereo-Bildpaare erzeugt werden, die mit geeigneten optischen Hilfsmitteln oder bei einiger Übung (5) auch mit bloßem Auge betrachtet werden können.
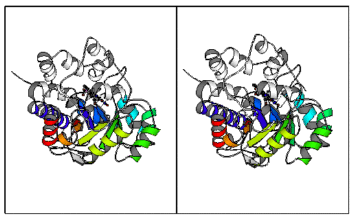
Hier das Script zur Darstellung des TMV-Proteins mit MolScript. Das Programm liest das Script zeilenweise; rechts von einem Ausrufezeichen stehen Kommentare für den Benutzer.
plot
read mol "2tmv.pdb"; ! PDB-Eintrag der TMV-Koordinaten
transform atom * by centre position in amino-acids
by rotation x 180.0; ! Positionierung des Moleküls
set planecolour yellow; ! Farbe für das Proteinband
set plane2colour white; ! Farbe der Innenseiten
set linewidth 2.0; ! Strichstärke kräftiger
set depthcue 1.0; ! Maß der Tiefenwirkung
coil from P1 to P9; ! Angabe der darzustellenden
helix from P9 to P14; ! Struktur im Proteinanteil
coil from P14 to P19;
helix from P19 to P33;
coil from P33 to P37;
helix from P37 to P52;
coil from P52 to P73;
helix from P73 to P86;
coil from P86 to P97;
turn from P97 to P100;
coil from P100 to P103;
turn from P103 to P106;
coil from P106 to P111;
helix from P111 to P135;
coil from P135 to P140;
helix from P140 to P146;
coil from P146 to P153;
set planecolour red; ! Farbe für die Basen GAA
set linecolour red;
set linewidth 3.0; ! besser sichtbare Linien
bonds in from R1 to R3; ! Bindungen zwischen allen Atomen
! sollen gezeigt werden
end_plot
Literatur: