Hier folgt eine Liste aller in tRNAPhe enthaltenen Nucleoside
 |
|
Adenosin (A, 5,9,13,14,21,23,29,31,35,36,38,39,44,62,64,66,67,73,76) in tRNA: | |
 |
|
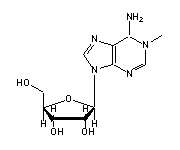
1-Methyladenosin (1MA, 58) in tRNA: | |
 |
|
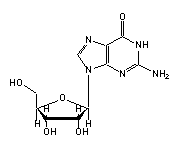
Guanosin (G, 1,3,4,15,18-20,22,24,30,42,43,45,51,53,57,65,71) in tRNA: | |
 |
|
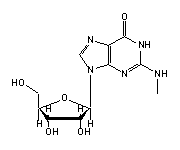
N2-Methylguanosin (2MG, 10) in tRNA: | |
 |
|
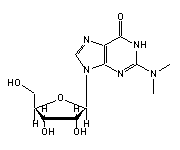
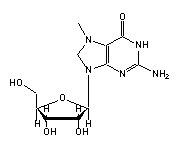
N2-Dimethylguanosin (M2G, 26) in tRNA: | |
 |
|
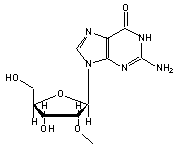
7-Methylguanosin (7MG, 46) in tRNA: | |
 |
|
O2'-Methylguanosin (OMG, 34) in tRNA: | |
 |
|
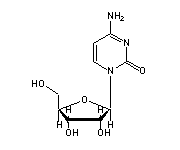
Cytidin (C, 2,11,13,25,27,28,48,56,60,61,63,70,72,74,75) in tRNA: | |
 |
|
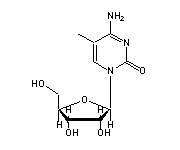
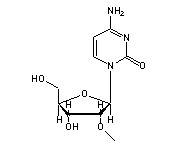
5-Methylcytidin (5MC, 40,49) in tRNA: | |
 |
|
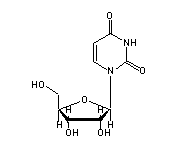
O2'-Methylcytidin (OMC, 32) in tRNA: | |
 |
|
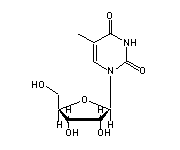
Uridin (U, 6-8,12,33,41,49,50,52,59,68,69) in tRNA: | |
 |
|
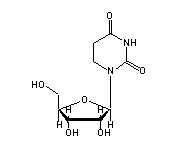
5-Methyluridin (5MU, 54) in tRNA: | |
 |
|
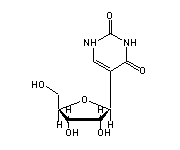
5,6-Dihydrouridin (H2U, 16,17) in tRNA: | |
 |
|
Pseudouridin (PSU, 39,55) in tRNA: | |
 |
|
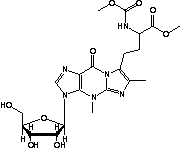
Wybutosin (YG, 37) in tRNA: | |
In tRNAPhe gepaarte Basen sind:
Ungepaarte Basen
Die 'Arm'-Bezeichnungen stammen aus der Zeit, als nur die Sekundärstruktur der tRNA bekannt war ('Kleeblatt'). In der Tertiarstruktur führt das allenfalls zu Verwirrung
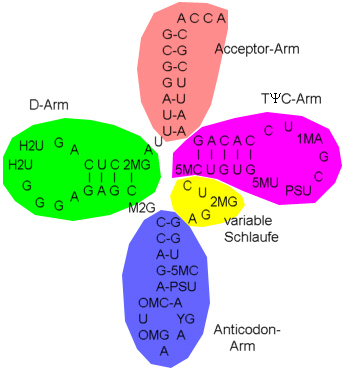
Die rot und dunkelgrün markierten Teile der Struktur
Die Ribose-Phosphat-Kette der tRNA wird teilweise durch Wassermoleküle stabilisiert, gezeigt hier am Beispiel des T psi C-Arms (G51 - U59)
Die Basen von D-Arm und T psi C-Arm sind fest aneinander gebunden, dieser zentrale Teil des Moleküls ist am starrsten aufgebaut
Je zwei Wassermoleküle verbinden über Wasserstoffbrücken die 2'-Hydroxylgruppe der Ribose mit dem Phosphat des nächsten Nucleotides, z. B.
H Shi & PB Moore, The crystal structure of yeast phenylalanine tRNA at 1.93 Å resolution: A classic structure revisited, RNA 6 (2000) 1091-1105
L Jovine et al, The crystal structure of yeast phenylalanine tRNA at 2.0 Å resolution: Cleavage by Mg2+ in 15-year old crystals, J. Mol. Biol. 301 (2000) 401-414
10-01 - R Bergmann